Ziel der molekulargenetischen Diagnostik ist es, Veränderungen der DNA in einzelnen Genen festzustellen bzw. auszuschließen, die lichtmikroskopisch nicht nachweisbar sind.
Voraussetzung dafür ist eine möglichst präzise Verdachtsdiagnose, die vom behandelnden Arzt oder im Rahmen einer genetischen Beratung gestellt wird. Bei klinischem Verdacht auf eine bekannte erbliche Erkrankung erfolgt dann die Suche nach der zugrunde liegenden genetischen Veränderung (Mutation) zielgerichtet im jeweiligen Gen. Entsprechend der Fragestellung wird die Mutation mit der jeweils geeigneten molekulargenetischen Technik dargestellt. Wir beraten Sie gerne, welches diagnostische Vorgehen im Einzelfall am sinnvollsten ist.
Der Nachweis einer ursächlichen Mutation bei einer erkrankten Person ermöglicht dann ggf. auch eine prädiktive (vorhersagende) genetische Diagnostik bei den gesunden Risikopersonen der betroffenen Familie zum sicheren Nachweis oder Ausschluss der Mutation. Hierdurch können die Risikopersonen frühzeitig erkannt und weitere Maßnahmen, wie z.B. Früherkennungs-Untersuchungen bei erblichen Tumorsyndromen eingeleitet werden.
Eine pränatale oder prädiktive genetische Diagnostik darf gemäß Gendiagnostikgesetz aufgrund der potenziell weitreichenden Konsequenzen eines genetischen Befundes nur nach vorheriger humangenetischer Beratung durchzugeführt werden.
In unserem molekulargenetischen Labor werden medizinisch-genetische Fragestellungen aus den folgenden Bereichen bearbeitet:
- Erbliche Tumorerkrankungen, zum Beispiel Mammakarzinom, Kolonkarzinom
- Hämatologie und Gerinnungsstörungen, zum Beispiel Alpha- und Beta- Thalassämie, Protein S- und Protein C- Defizienz
- Neurogenetik und mentale Retardierung, zum Beispiel Fragiles X-Syndrom, Tuberöse Sklerose, Neurofibromatose
- Reproduktionsgenetik, zum Beispiel AZF-Deletion, CBAVD
- Erbliche Stoffwechselerkrankungen, zum Beispiel die hereditäre Pankreatitis, das Mittelmeerfieber und andere hereditäre Fiebersyndrome, MODY-Diabetes, Mukoviszidose, Morbus Wilson
- Syndromdiagnostik, zum Beispiel Di-George Syndrom, Noonan-Syndrom, SHOX-Haploinsuffizienz
- Bindegewebserkrankungen, zum Beispiel Marfan-Syndrom, Ehlers-Danlos-Syndrom
- Herzerkrankungen, zum Beispiel Kardiomyopathien, Brugada-Syndrom, Long-QT-Syndrom
- HLA-Typisierung, zum Beispiel Narkolepsie, Morbus Behçet
Das vollständige Anforderungsformular inkl. aller Genanalysen finden Sie hier zum Download.
Für weitere Fragen zur molekulargenetischen Diagnostik (z.B. Rückfragen zu individuellen Panels, Bearbeitungszeiten, Probenmaterial etc.) stehen wir Ihnen sehr gerne zur Verfügung.
Sie können uns telefonisch über unsere Zentrale 0941-94 6822-0 oder per Email unter genetik@labor-staber.de erreichen.
MOLEKULARGENETISCHE TECHNIKEN:
Next Generation Sequencing
Next Generation Sequencing (NGS) ist ein Hochdurchsatzsequenzier-Verfahren, welches unter anderem für die parallele Sequenzierung mehrerer Gene (Gen-Panel-Analyse), die parallele Sequenzierung eines Großteils der kodierenden Abschnitte (Whole-Exome-Analyse) oder die Sequenzierung des gesamten Genoms (Whole-Genome-Analyse) eingesetzt wird. Je nach Fragestellung werden vor der eigentlichen Sequenzierung bestimmte Bereiche des Genoms durch Hybridisierung mit spezifischen Sonden oder durch gezielte Amplifikation mittels Multiplex-PCR angereichert. Die so erzeugten doppelsträngigen DNA-Fragmente werden mit Adaptern, Indices und gegebenenfalls molekularen Tags versehen, wodurch eine eindeutige Zuordnung jedes einzelnen DNA-Moleküls gewährleistet wird. Nach der Denaturierung und der Sequenzierung dieser DNA-Bibliotheken werden die erhaltenen Sequenzdaten bioinformatisch sortiert, gefiltert und die Sequenzen jedes Einzelmoleküls individuell an der Referenzsequenz des humanen Genoms ausgerichtet. So entsteht ein Datensatz, der in der Regel jeden der zu sequenzierenden Bereiche mit mehreren Einzelsequenzen abdeckt. Die Höhe dieser Abdeckung (= Coverage) der zu analysierenden Gene bestimmt die Qualitätsstufe der Diagnostik: durch höhere Coverage-Werte können Lesefehler ausgeschlossen und Punktmutationen nachgewiesen werden. Die Datensätze enthalten darüber hinaus Hinweise auf Deletionen oder Duplikationen (Copy Number Variations, CNVs) einzelner Exons.
Strukturelle Chromosomenanomalien, Repeatexpansionen (z. B. Fragiles-X-Syndrom) und Varianten in Bereichen mit Homopolymeren oder Pseudogenen werden durch die meisten DNA-Sequenzierungsmethoden beim derzeitigen Stand der Technik nicht zuverlässig erfasst. In diesen Fällen werden bei Bedarf alternative Methoden eingesetzt (z. B. Long-Range-PCR, Array-CGH, Southern Blot).
PCR
Die Polymerase-Kettenreaktion (PCR) ist eine Methode zur Vervielfältigung (Amplifikation) von DNA. Die zu amplifizierende Zielsequenz wird durch die Sequenz zweier synthetischer einzelsträngiger DNA-Oligonukleotide definiert, die aufgrund ihrer Basenpaarabfolge nach dem Aufschmelzen des doppelsträngigen DNA-Moleküls (Denaturierung) spezifisch mit flankierenden Sequenzen auf dem Sense- beziehungsweise dem Antisense-Strang antiparallel hybridisieren (Annealing). Die hybridisierten Oligonukleotide mit ihren freien 3'-Enden dienen als Primer der DNA-Polymerase, die in der Folge in Sense- und Antisense-Richtung durch den Einbau von Desoxynukleotiden (dNTPs) jeweils einen neuen Gegenstrang synthetisiert (Elongation). Nach wiederholten Zyklen von Denaturierung, Annealing und Elongation dienen fast ausschließlich neu synthetisierte DNA-Stränge als Vorlagen für die nachfolgenden Zyklen. Dies hat zur Folge, dass nur der von beiden Oligonukleotiden flankierte DNA-Bereich unter allen PCR-Produkten exponentiell angereichert wird. Die PCR ermöglicht so eine Analyse der Zielsequenz auch bei geringen Probenmengen und ist der Ausgangspunkt für viele weitere Anwendungen wie die DNA-Sequenzierung nach Sanger oder gentechnologische Verfahren wie die Klonierung von Genen.
Durch zahlreiche Erweiterungen und Verbesserungen sind auch hochspezialisierte Variationen der Methode wie beispielsweise die quantitative Echtzeit-PCR (qPCR, real-time PCR) oder die Multiplex-PCR (z. B. MLPA) im Laboralltag im Einsatz.
Agarosegelelektrophorese
Die Agarosegelelektrophorese ist eine Methode, mittels derer Nukleinsäuren (z. B. PCR-Produkte) durch Anlegen eines elektrischen Feldes auf einem Agarosegel aufgetrennt werden. Durch Zugabe interkalierender Fluoreszenzfarbstoffe (z.B. Ethidiumbromid) werden die Nukleinsäuren unter UV-Licht detektiert. Da kürzere Moleküle schneller durch die Gelmatrix wandern als längere, kann mit dieser Technik zum Beispiel die Größe eines zu analysierenden DNA-Moleküls durch den Vergleich mit einer mitlaufenden Standard-DNA bestimmt werden.
Kapillarelektrophorese
Die Kapillarelektrophorese ist eine weitere Methode, die zur Auftrennung von DNA-Molekülen eingesetzt wird. Hier findet die Auftrennung in einem dünnen Kapillarrohr, das mit einer Gelmatrix befüllt ist, statt. Die Probe wird elektrokinetisch injiziert und durch das Anlegen einer Spannung im Kilovoltbereich beginnt die elektrophoretische Wanderung und damit die Auftrennung der DNA entlang der Kapillare. Die einzelnen DNA-Fragmente sammeln sich in verschiedenen Zonen und werden an einem Detektor vorbeigetrieben, der das Passieren eines Fragments registriert und als Peak-Signal aufzeichnet. Die Kapillarelektrophorese ermöglicht im Vergleich zur Agarosegelelektrophorese eine wesentlich höhere Auflösung und wird beispielsweise bei der Fragmentlängenanalyse und bei der Sequenzierung nach Sanger eingesetzt.
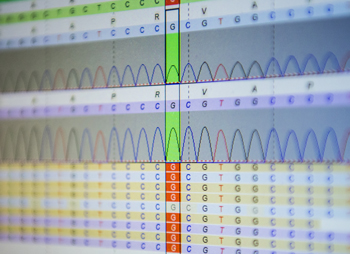
DNA-Sequenzierung nach Sanger
Mit der DNA-Sequenzierung nach Sanger wird die Abfolge der Basen eines DNA-Moleküls (z.B. eines PCR-Produkts) bestimmt. Der Startpunkt der Sequenzreaktion wird durch ein einzelsträngiges synthetisches Oligonukleotid definiert, welches aufgrund seiner Sequenz mit einer bestimmten Region auf dem Antisense-Strang des zu sequenzierenden DNA-Moleküls hybridisieren kann. Nach dem Aufschmelzen des Templates dient das Oligonukleotid mit seinem freien 3'-Ende als Primer für die DNA-Polymerase. Diese verlängert den Sense-Strang in einer isothermen Reaktion komplementär zum Antisense-Strang durch den sukzessiven Einbau freier Desoxynukleotide (dNTPs). Da der Reaktionslösung in einem bestimmten Verhältnis vier verschiedene Fluorophor-gekoppelte Didesoxynukleotide (ddNTPs, Abbruchnukleotide) beigemischt sind, entstehen unterschiedlich lange Teil-Kopien des Templates, die am 3'-Ende mit einem der letzten Base entsprechenden Fluorophor markiert sind.
Die anschließende Kapillarelektrophorese ermöglicht die Auftrennung der Teil-Kopien mit einer Auflösung von einem Basenpaar. Durch Anregung der Fluorophore mit einem Laser kann das Sequenziergerät anhand der emittierten Signale der am Detektor vorübermigrierenden Fragmente jeder Position in Echtzeit eine korrespondierende Base zuordnen. Am Ende der bioinfomatischen Aufbereitung der Rohdaten steht eine lesbare Sequenz, welche die Identifizierung von Punktmutationen oder kleinen Deletionen bzw. Duplikationen ermöglicht.
Fragmentlängenanalyse
Bei der Fragmentlängenanalyse werden PCR-Produkte, die mit fluoreszenzmarkierten Primern erzeugt wurden, mittels Kapillarelektrophorese aufgetrennt und detektiert. Anhand einer Eichkurve, die aus dem Laufverhalten eines bekannten Standards ermittelt wird, können die Fragmentlängen der DNA aus der PCR-Reaktion berechnet werden. Dies wird beispielsweise für die Diagnostik von Repeatexpansionen wie beim Fragilen-X-Syndrom, die Deletions- und Duplikationsanalyse mittels MLPA oder auch Vaterschaftsanalysen eingesetzt.
MLPA
Die MLPA (Multiplex Ligation-dependent Probe Amplification) erlaubt die gleichzeitige Analyse von definierten DNA-Abschnitten (Loci) im Genom im Hinblick auf Kopiezahlveränderungen (Deletionen oder Duplikationen) einzelner Genbereiche oder ganzer Gene. Das Verfahren beruht auf einer Hybridisierung von spezifischen Oligonukleotiden, die jeweils paarweise an benachbarte Nukleotide der Zielsequenz binden können und anschließend über eine Ligation miteinander verknüpft werden. Die Menge der sequenzspezifischen Ligationsprodukte ist dabei proportional zur Kopiezahl der entsprechenden Ziel-Sequenz. Nach einer PCR mit einem Fluoreszenz-markierten Primer werden die Amplifikationsprodukte durch Kapillarelektrophorese der Größe nach getrennt. Durch einen Vergleich der erhaltenen Peakflächen für jede Zielsequenz des Patienten und parallel untersuchter Kontrollpersonen werden Dosisunterschiede erkennbar, wenn an der entsprechenden Stelle in der DNA des Patienten eine Deletion oder Duplikation vorliegt.
Southern Blot
Das Southern-Blot-Hybridisierungsverfahren ermöglicht den Nachweis einer spezifischen Sequenz in einem Genom. Dafür wird die genomische DNA durch teils methylierungssensitive Enzyme (Restriktionsendonukleasen) verdaut und durch eine Gelelektrophorese aufgetrennt. Nach einer alkalischen Denaturierung werden die nun als Einzelstränge vorliegenden DNA-Fragmente auf eine Membran übertragen und dort fixiert. Durch Zugabe einer radioaktiv oder chemisch markierten RNA-Gensonde, die an die gesuchte Sequenz bindet kann die Zielsequenz auf der Membran nachgewiesen werden. Die Methode wird eingesetzt, um beispielsweise sehr große Repeatexpansionen wie Vollmutationen beim Fragilen-X-Syndrom nachzuweisen.
